使用 PCA 进行降维可视化,了解特征分布
降维是数据挖掘流程中,一种对高维度特征数据预处理方法。降维是将高维度的数据保留下最重要的一些特征,去除噪声和不重要的特征,从而实现提升数据处理速度的目的。在实际的生产和应用中,降维在一定的信息损失范围内,可以节省大量的时间成本。降维也成为应用非常广泛的数据预处理方法。另外,除了让算法运行更快,效果更好。降维还有一种场景,就是将数据可视化,根据数据分布情况进而选择合适算法。
降维算法主要有:主成分分析(PCA)、奇异值分解(SVD)、因子分析(FA)、独立成分分析(ICA)。本篇文章主要介绍利用 PCA 进行可视化。
在 sklearn 库,PCA 在 sklearn.decomposition.PCA 类:
class sklearn.decomposition.PCA(n_components=None, copy=True, whiten=False, svd_solver=’auto’, tol=0.0,iterated_power=’auto’, random_state=None)
案例说明
# 导入相关库
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
from sklearn.decomposition import PCA
# 获取skearn库鸢尾花数据集,鸢尾花四维数组
iris = load_iris()
y = iris.target
X = iris.data
# 查看原始数据
X[0:5] array([[5.1, 3.5, 1.4, 0.2],
[4.9, 3. , 1.4, 0.2],
[4.7, 3.2, 1.3, 0.2],
[4.6, 3.1, 1.5, 0.2],
[5. , 3.6, 1.4, 0.2]])# 调用PCA,n_components=2
pca = PCA(n_components=2)
pca = pca.fit(X)
X_dr = pca.transform(X)
# 查看降维后的数据
X_dr[0:5]array([[-2.68412563, 0.31939725],
[-2.71414169, -0.17700123],
[-2.88899057, -0.14494943],
[-2.74534286, -0.31829898],
[-2.72871654, 0.32675451]])# 鸢尾花三种类型可视化
colors = ['red', 'black', 'orange']
iris.target_names
plt.figure()
for i in [0, 1, 2]:
plt.scatter(X_dr[y == i, 0] ,X_dr[y == i, 1]
,alpha=.7
,c=colors[i] ,label=iris.target_names[i])
plt.legend()
plt.title('PCA of IRIS dataset')
plt.show()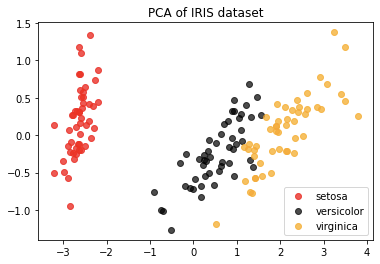
如图,降维后进行可视化,可以清楚看到鸢尾花数据集 3 种类型分布,观察分布可以得知,鸢尾花数据集使用聚类算法,应该会取得比较好的效果。